Molekulare Spurensuche
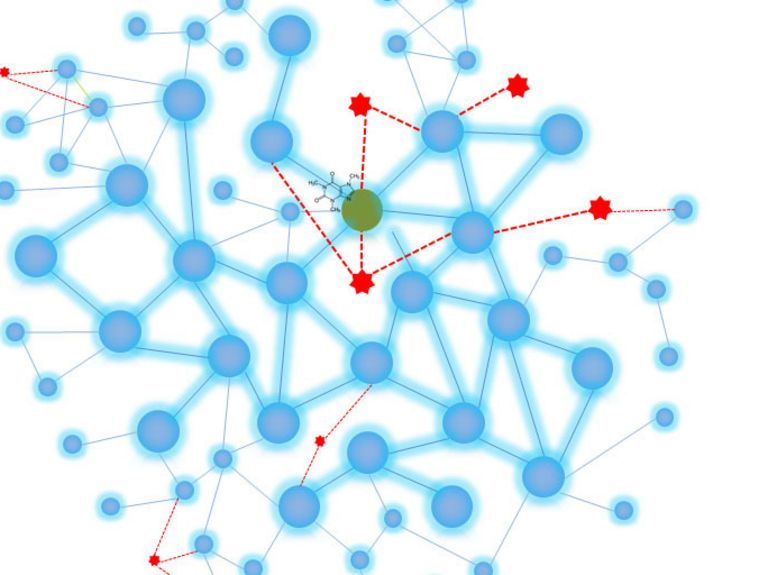
Schematische Darstellung des menschlichen Protein-Netzwerks (blau). Virale Proteine und deren Interaktionen mit den menschlichen Proteinen in rot. Grafik: Pascal Falter-Braun
Forscher vom Helmholtz Zentrum München untersuchen in einem von der EU geförderten Projekt, ob sich schon zugelassene Medikamente für die Behandlung von COVID-19 eignen. Um auf die richtige Spur zu kommen, versuchen sie zu verstehen, welche Proteine, Signalwege und molekularen Strukturen das Virus im menschlichen Körper ausnutzt und verändert.
Pascal Falter-Braun vom Helmholtz Zentrum München, Deutsches Forschungszentrum für Gesundheit und Umwelt (HMGU), koordiniert das EU-Projekt RiPCoN („Rapid interaction profiling of 2019 nCoV for network based deep drug repurpose learning“). Gemeinsam mit Partnern aus Frankreich und Spanien erforscht sein Team die molekularen Wechselwirkungen zwischen Virus und Mensch.
Herr Falter-Braun, Sie sind Direktor des Instituts für Netzwerkbiologie am HMGU. Viren standen dort bislang weniger im Fokus. Was ist ihr Ausgangspunkt?
Wir erforschen, wie die molekularen Netzwerke innerhalb einer Zelle funktionieren. Das Geschehen dort ist sehr komplex: Die vielen Proteine, Eiweißstoffe also, arbeiten nicht alleine, sondern erfüllen ihre Aufgaben immer gemeinsam mit anderen Proteinen in einem Netzwerk. Eine wichtige Aufgabe ist die Verarbeitung von Signalen, zum Beispiel bei Prozessen, die die Zellvermehrung regulieren, welche mal beschleunigt, mal gedrosselt werden muss – oder aber auch bei Immunantworten. Faszinierend und wichtig ist, dass diese Netzwerke im Prinzip ähnlich aufgebaut sind, wie soziale oder andere komplexe Netzwerke. Einige Proteine sind sehr stark vernetzt, wie ein ‚Influencer‘ oder wichtiger Flughafenknotenpunkt – andere Proteine finden sich eher am Rande und sind sehr auf eine eigenständige Aufgabe fokussiert.
Wie nutzen Sie nun dieses Wissen?
Alle Viren dringen in menschliche Zelle ein und vermehren sich dort. Auch beim neuartigen Coronavirus werden dann Viren-eigene Eiweißstoffe hergestellt, die wiederum mit dem zelleigenen Protein-Netzwerk des Wirtes, also des Menschen, interagieren und versuchen es umzuprogrammieren. Um genau zu verstehen, was das Virus macht, schauen wir uns dieses Zusammenspiel auf Ebene der Proteine an. Welche Faktoren beeinflussen es? Genau dort setzt unser Projekt an.
Wie gehen Sie dabei vor? Sie selbst sind derzeit im Home Office...
Die erste Phase der Laborarbeiten läuft schon seit mehreren Wochen, trotz herausforderndem Minimalbetrieb. Wir haben Gen-Abschnitte des Virus synthetisieren lassen und überprüfen nun deren Interaktion mit fast allen menschlichen Proteinen – also um die 18.000 Stück. Das machen wir übrigens für alle humanen Coronaviren, also nicht nur für SARS-CoV-2, sondern auch für MERS, das SARS-Virus von 2003 und vier weitere, harmlosere. So vertiefen wir unser Wissen über die ganze Virenklasse.
Pascal Falter-Braun leitet das Institut für Netzwerkbiologie am Helmholtz Zentrum München (HMGU) und ist Professor für Mikroben-Wirts-Interaktion an der Ludwig-Maximilians-Universität München (LMU). Bild: HMGU
Sie nutzen dafür ein hefebasiertes System. Wie genau funktioniert das?
Wir nutzen einen besonderen Trick. Sehr vereinfacht dargestellt sorgen wir dafür, dass durch gentechnische Methoden in einer Hefezelle je ein virales und ein menschliches Protein hergestellt werden. Diese Hefezellen sind nun so präpariert, dass sie nur weiterleben und sich teilen, wenn die menschlichen und viralen Proteine aneinander binden. Die Bindung zeigt, dass die Proteine auf molekularer Ebene miteinander interagieren. Durch Sequenzierung können wir dann nachvollziehen, bei welchen der Millionen getesteten Paaren eine Bindung eintrat.
Was machen Sie dann mit dieser Information?
Wir haben den Vorteil, dass wir das Protein-Interaktions-Netzwerk im Menschen bereits sehr gut kennen und von sehr vielen menschlichen Proteine wissen, an welchen Aufgaben sie beteiligt sind. Das lässt sich auch gut auf Netzwerkkarten, ähnlich einem U-Bahn-Plan, darstellen. Jetzt wollen wir prüfen, in welche dieser Prozesse die viralen Proteine eingreifen, welche Netzwerke in der Nachbarschaft liegen und vor allem auch, an welchen Krankheiten die von den Viren angegriffenen Prozesse Anteil haben. Wir wollen wissen, wie sich die durch das Virus gestörten Prozesse korrigieren lassen, um so einen Weg zu finden, um die Vermehrung des Virus zu verhindern.
Kommen hier also Untersuchungen zu Medikamenten ins Spiel?
Ja. Im nächsten Schritt wollen wir untersuchen, welche bekannten Medikamente das Netzwerk bereits an diesen Stellen beeinflussen und so eventuell auch geeignet wären, SARS-CoV-2 zu neutralisieren. Das würde die zeitaufwändigen Zulassungswege ersparen oder abkürzen. Eine hohe Treffer-Wahrscheinlichkeit haben per se Medikamente, die bereits gegen Viren wirken. Dazu laufen natürlich auch anderswo viele Experimente. Unsere Nische ist, dass wir weniger naheliegende Präparate ausfindig machen können.
…wobei Sie sich durch künstliche Intelligenz und maschinelles Lernen unterstützen lassen.
Mit den gewonnenen Informationen können wir mit Hilfe von Computermodellen herausfinden, wie hoch die Erfolgsaussichten einzelner Medikamente sind. Hier werden wir wirklich alle zugelassenen Medikamente in den Blick nehmen. Die Unterstützung durch KI ist bei diesen riesigen Datenmengen enorm wichtig. An welchen Punkten im Netzwerk wirken sich sowohl Virus, Krankheiten und Medikamente aus? All solche Informationen können wir dank der Partner in Frankreich und Spanien kombinieren. Unsere Partner in Frankreich haben außerdem Modelle entwickelt, mit denen sich Wechselwirkungen von Proteinen und RNA-Molekülen vorhersagen lassen. Das ist wichtig, da Coronaviren als erstes immer ein RNA-Molekül in die menschliche Wirtszelle einschleusen.
Wäre das Projekt mit seinen Modellierungen vor 5 Jahren bereits so möglich gewesen?
Nein beziehungsweise nur in Teilen deren Aussagekraft dramatisch geringer gewesen wäre.
Wie schnell rechnen Sie mit Ergebnissen?
Das Erstellen der Netzwerkkarten und die weiteren Analysen durch die Partner werden etwas Zeit brauchen. Ich hoffe, dass wir in drei bis vier Monaten die ersten Kandidaten haben, mit denen wir in-vitro-Experimente machen können.
Spüren Sie denn, auch in Anbetracht der Dringlichkeit, einen gewissen Erwartungsdruck?
Den macht man sich eher selbst, denn mit solch einer Projektbewilligung fühlen wir uns natürlich in der Pflicht. Gleichzeitig ist die Aussicht, in dieser Zeit sinnvolles beisteuern zu können, enorm motivierend. Und: Wir werden in jedem Fall Beiträge zu dem Verständnis der Corona-Viren leisten können – selbst, wenn wir am Ende kein „Medikament abliefern“, wovon wir aber nicht ausgehen.
Es ist noch eine zweite Projektphase geplant. Sie wollen herausfinden, welche Auswirkungen natürlich vorkommende genetische Unterschiede der interagierenden menschlichen und viralen Proteine auf den individuellen Krankheitsverlauf haben. Wie lässt sich die Idee dahinter erklären?
Wir alle tragen leicht unterschiedliche genetische Information für viele unserer Proteine in uns. Das führt unter anderem dazu, dass manche von uns für manche Krankheiten – von Allergien über Krebs bis zu Erbkrankheiten – anfälliger sind. Es ist sehr stark anzunehmen, dass auch die viralen Proteine mit den Proteinen von manchen Menschen, abhängig von der Ausformung, leichter wechselwirken können. Je nachdem, wo diese „bessere Interaktion“ auftritt, könnte das zu unterschiedlichen Krankheitsverläufen führen: zu schwereren, aber auch zu milderen, wenn es etwa um Proteine geht, die das Virus besser abfangen können. Die zentrale Frage ist also: Was sind die Zielproteine des Virus und in welchen Varianten gibt es die beim Menschen? In der weiteren Analyse wollen wir unsere Antworten darauf mit echten Krankheitsdaten zusammenbringen und daraus Rückschlüssen für andere Patienten ziehen. Auch damit wollen wir möglichst schnell beginnen. Das ist aufwändig und wir werden auf Daten aus anderen Forschungsprojekten zurückgreifen müssen. Aber ich bin zuversichtlich, dass sich das lohnt, denn die Erkenntnisse daraus ließen sich für zukünftiges Risikomanagement und die Ressourcenplanung von Kliniken nutzen.
Vielen Dank für das Gespräch!
Aktuelle Forschung, Zahlen und Fakten zu SARS-CoV-2
Hier finden Sie Erklärungen wichtiger Begriffe rund um das Coronavirus SARS-CoV-2.
Leser:innenkommentare