Interview
„KI wird ein Gamechanger in der Antibiotikaforschung“
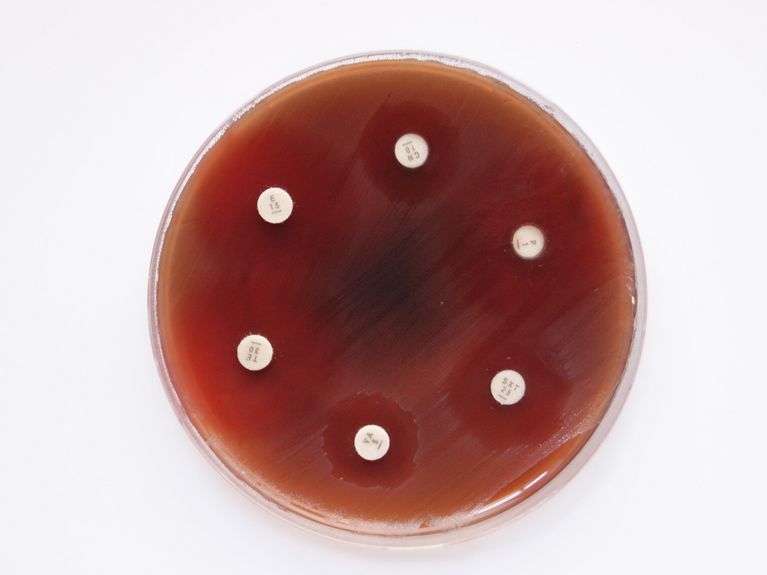
Das Bild zeigt einen Hemmhoftest, mit dem überprüft wird, ob Bakterien gegen bestimmte Antibiotika resistent sind. Bild: HZI/René Bergmann, Patric Nitsche-Schmitz
Bereits heute sterben jährlich 700.000 Menschen wegen multiresistenter Keime, Tendenz steigend. Wie Künstliche Intelligenz dabei helfen kann, neue Antibiotika zu entwickeln, erklärt Andreas Keller vom Helmholtz-Institut für Pharmazeutische Forschung Saarland (HIPS).
Herr Professor Keller, Antibiotikaresistente Keime werden immer mehr zum Problem – warum ist das so?
Antibiotika wurden in den letzten Jahrzehnten sehr viel eingesetzt, zu viel: Nicht nur beim Menschen werden sie zu häufig verschrieben, auch in Fällen, in denen zum Beispiel nur eine Virusinfektion vorliegt und sie unwirksam sind. Auch beim Tier, in den Mastbetrieben, werden viele Antibiotika eingesetzt. Der viele Kontakt der Bakterien mit Antibiotika führt dazu, dass sich sogenannte Multiresistenzen entwickeln: Bakterien werden immer widerstandsfähiger gegen die weit verbreiteten Medikamente. Schon heute sterben jährlich weltweit 700.000 Menschen wegen multiresistenter Keime, schätzen Experten. Bis 2050 könnte diese Zahl Prognosen zufolge auf 10 Millionen steigen. Doch eigentlich könnten wir das verhindern, und hier kommen wir zu den Möglichkeiten: Wir könnten mit neuen Antibiotika aufrüsten. Denn in den letzten Jahren wurden in zehntausenden Proben aus dem Umfeld von Menschen, Tieren oder aus der Umwelt durch Deep Sequencing genetisch entschlüsselt. Dabei wurden etliche tausend potenzielle neue Naturstoffe gefunden, die sicherlich auch Antibiotika-Kandidaten beinhalten …
… die auch gegen multiresistente Keime wirken?
Ja, davon ist auszugehen. Allerdings wurden in den letzten Jahren die Forschungsinvestitionen vor allem von Seiten der Pharmakonzerne stark zurückgefahren. Mit Zunahme der Resistenzen könnte das tatsächlich auf eine medizinische Katastrophe hinauslaufen.
Warum investieren die Firmen nicht in den Bereich, wenn der Bedarf so groß ist?
Das hat verschiedene Gründe, die zusammenkommen: Einerseits sind Antibiotika wenig einträglich, denn in der Regel werden sie nur für ein paar Tage gegeben, dann sind die Erreger besiegt und sie werden nicht mehr gebraucht. Im Vergleich zu Medikamenten gegen chronische Krankheiten lässt sich mit Antibiotika eher wenig Geld verdienen. Hinzu kommt der Entwicklungsaufwand. Alleine unsere tausenden neuen Naturstoffe herzustellen und auf Wirksamkeit zu testen ist mit herkömmlichen Methoden fast unmöglich.
Also keine Besserung in Sicht?
Professor Dr. Andreas Keller leitet am Helmholtz-Institut für Pharmazeutische Forschung Saarland (HIPS) in Saarbrücken die Abteilung Klinische Bioinformatik (CLIB), außerdem hat er an der Universität er des Saarlandes eine Professur Klinische Bioinformatik inne. Bild: Oliver Dietze / HIPS
Ich bin überzeugt, dass KI ein Gamechanger in der Antibiotikaforschung werden wird. Damit lässt sich die Suche nach den wirksamen Antibiotika unter den Naturstoffen enorm beschleunigen und damit die Entwicklung billiger und schneller machen. Das HIPS, und die Universität des Saarlandes haben das Projekt NextAID ins Leben gerufen, das ist die die Abkürzung für „Neuro-explicit AI for Drug Discovery“. Inzwischen kooperieren wir unter anderem auch mit dem Helmholtz-Zentrum für Informationssicherheit, kurz CISPA, gestern erst haben mein geschätzter Kollege Jilles Vreeken vom CISPA und ich uns wieder getroffen dazu.
Wie genau hilft Künstliche Intelligenz bei der Suche nach den passenden Antibiotika?
Zurzeit hilft sie vor allem beim Priorisieren, und das ist schon ein entscheidender Vorteil. In der Praxis funktioniert das so: Wir füttern die KI mit Daten über die Keime, ihre genetischen Eigenschaften, molekularbiologische Wirkungen, die Oberflächenstruktur der Zellen und wie sie auf verschiedene bekannte Antibiotika reagieren. Die Eigenschaften der Antibiotika „kennt“ die KI ebenfalls. Dann berechnet sie daraus die Wahrscheinlichkeiten, welche Abwehrmechanismen zu Resistenzen führen und welche Naturstoffe am ehesten Fähig sind, diese zu umgehen oder zu durchbrechen. Dann bekommen wir vom Computer zum Beispiel eine Top-20-Liste derjenigen Naturstoffe, die nach KI-Berechnungen am wahrscheinlichsten geeignet sind, bestimmte multiresistente Keime zu knacken.
Damit die KI hier tatsächlich vielversprechende Substanzen in die Liste aufnimmt, muss sie ja zunächst mit einigen Daten gefüttert werden. Müssen diese Daten nicht auch erst erhoben werden, oder zumindest in eine Form gebracht werden, dass die KI damit etwas anfangen kann?
Es stimmt, grundsätzlich gilt: Je mehr Daten wir der KI – in der richtigen Qualität natürlich – geben, desto besser wird ihre Priorisierung. Hier kommt uns eine Entwicklung in der internationalen Forschung entgegen: Der Trend geht stark hin zu mehr Datenaustausch nach dem sogenannten FAIR-Prinzip, das steht für Findable, Accessible, Interoperable und Reusable. Zusätzlich wird Federated Learning eingesetzt: Dabei trainieren verschiedene Einrichtungen KI Modelle mit ihren Daten und helfen so dabei, ein robusteres Modell zu erstellen. Klar, das Sammeln der Daten und das Trainieren dauert auch erst einmal ein bis zwei Jahre. Aber selbst wenn man diese Zeit mit einberechnet, kann KI trotzdem noch eine enorme Zeitersparnis bringen.
An Antibiotika gegen welche Erreger arbeiten Sie aktuell bei NextAID und im HIPS?
Einer unserer Schwerpunkte ist die Behandlung von Tuberkulose. Das ist global betrachtet ein riesiges Problem: WHO-Schätzungen zufolge erkranken jährlich mehr als zehn Millionen Menschen, 1,6 Millionen von ihnen sterben daran. Man könnte in Deutschland meinen, das ist weit weg, aber angesichts des globalen Reiseverkehrs, der Migrationsströme und des Klimawandels wird Tuberkulose sich auch wie der in Deutschland ausbreiten. Und Resistenzen gegen die bewährten Kombinationstherapien aus verschiedenen Antibiotika treten leider immer häufiger auf.
Wie geht man weiter vor, wenn die KI zum Beispiel eine Liste der vielversprechendsten Substanzen zur Behandlung von Tuberkulose erstellt hat?
Zunächst lassen wir die Wirkstoffe produzieren. Das passiert biotechnologisch in den allermeisten Fällen mit Bakterien oder einzelligen Pilzen, wie zum Beispiel Hefen. Und dann probieren wir im Labor aus, wie gut sie jeweils gegen Tuberkulose-Erreger wirken.
Das klingt ja eigentlich recht einfach.
Vom Prinzip her ist es das auch, aber in der Praxis ist es dann doch komplizierter. Zunächst ist es nicht immer so, dass direkt unter den 20 Naturstoffen auch tatsächlich etwas Wirksames ist. Und dann muss man natürlich auch schauen, ob die Substanz Menschen überhaupt verabreicht werden kann. Um optimale Ergebnisse zu erzielen ist es wichtig, dass wir die KI mit möglichst vielen Daten neuer Naturstoffe füttern und selbst auch nach neuen Substanzen suchen. Dazu hat das HIPS zusammen mit der Naturlandstiftung Saar ein Community Science-Projekt namens MICROBELIX ins Leben gerufen: Dabei kann uns jede:r Bürger:in Bodenproben zum Beispiel aus dem eigenen Garten schicken, die dann im Labor analysiert werden auf darin enthaltene Naturstoffe. Eine weitere Quelle für neue Substanzen trägt jeder von uns im Darm: Das Mikrobiom.
Ist das so zu verstehen, dass in unserem Darm auch Antibiotika produziert werden?
Im Prinzip ja, Die Bakterien im Verdauungssystem produzieren eine Reihe von teilweise noch unbekannten Substanzen, die teilweise gegen Bakterien wirken, die nicht zum Mikrobiom gehören. Entsprechend können wir gezielter als sonst nach vielversprechenden Wirkstoffen fahnden. Wir fragen uns zum Beispiel: Warum ist ausgerechnet dieser und jener Patient weniger anfällig für bestimmte Erkrankungen? Wir suchen nach Antworten im Mikrobiom, also nach bestimmten Bakterienstämmen im Darm, die bei den „robusten“ Menschen vermehrt vorkommen, und dann schauen wir uns an, welche Substanzen diese Stämme produzieren. Solche Daten und Informationen lassen wir auch in die KI einfließen.
Und die Priorisierungslisten der KI verbessert sich weiter.
Ja, das hoffen wir natürlich. Womöglich sind solche Priorisierungslisten auch nur der Anfang der Möglichkeiten, die KI uns bei der Suche nach neuen Antibiotika bietet. Vielleicht wird KI bald schon so weit sein, dass sie uns bestimmte Molekülstrukturen vorschlägt als Kandidaten für wirksame Antibiotika – und wir versuchen dann, diese Strukturen zu designen. Man würde dann von generativer KI sprechen, also KI, die selbst etwas Neues vorschlägt. Vielleicht kommt das schon in ein oder zwei Jahren, das ist schwer vorherzusagen, möglich wäre es! Aber selbst heute leistet KI schon einen wesentlichen Beitrag dazu, dass die Menschheit beim Kampf gegen multiresistente Keime nicht den Anschluss verliert.
Die Forscher:innen am Helmholtz-Instituts für Pharmazeutische Forschung Saarland (HIPS) suchen nach neuen Therapein und Arzneistoffen gegen Infektionkrankheiten. Partner im HIPS sind das Helmholtz-Zentrum für Infektionsforschung (HZI) Braunschweig und die Universität des Saarlandes.
Das ebenfalls in Saarbrücken beheimatete Helmholtz-Zentrum für Informationssicherheit - CISPA ist an dem Projekt „Neuro-explicit AI for Drug Discovery“ beteiligt, in dem die Forscher:innen neue Antibiotioka mit Hilfe von KI entwickeln wollen.
Leser:innenkommentare