Selbstlernende Algorithmen
Wie KI die Bildauswertung revolutioniert
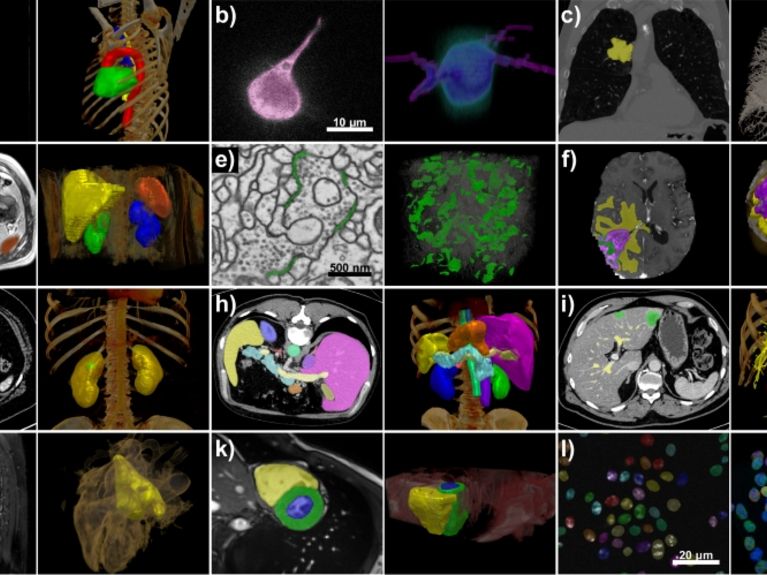
Das von Klaus Maier-Hein und seinem Team entwickelte Programm nnU-Net verarbeitet eine Vielzahl von Datensätzen und Bildeigenschaften. Alle Beispiele stammen aus den Testsätzen verschiedener internationaler Segmentierungs-Wettbewerbe. Bild: Isensee et al. / Nature Methods
Die Bildanalyse mit künstlicher Intelligenz (KI) ist in der Medizin ein großes Zukunftsthema. Medizininformatiker aus Heidelberg haben nun eine Methode entwickelt, mit der sich sämtliche Bilddaten mit KI bearbeiten lassen – kostenlos und auch für Laien nutzbar.
Der Medizininformatiker Fabian Isensee arbeitet in der Abteilung Medizinische Bildverarbeitung am Deutschen Krebsforschungszentrum DKFZ. Bild: DKFZ.
Was ist dort auf dem Bildschirm zu sehen? Ist das ein Teil eines Lungenkarzinoms? Oder gesundes Lungengewebe? Ein Gefäß? Ein Ausschnitt einer Faser des Herzmuskels? Mehrere Millionen Mal pro Minute stellt der Algorithmus, den der Medizininformatiker Fabian Isensee im Team von Klaus Maier-Hein am Deutschen Krebsforschungszentrum (DKFZ) nach einer neuen Methode hat trainieren lassen, diese Frage: Was ist hier zu sehen?
Für jeden Bildpunkt einer Aufnahme aus dem Körper eines Patienten, zum Beispiel ein MRT- oder CT-Bild, sucht und findet das Programm eine Antwort. Diese Unterscheidung, welcher Bildpunkt zu welcher Struktur gehört, wird in der Forschung zur künstlichen Intelligenz (KI) semantische Segmentierung genannt.
„In der Strahlentherapie markieren die Ärzte meist selbst mühsam das Krebsgewebe und die umgebenden Strukturen, das dauert oft mehrere Stunden“, sagt Fabian Isensee. „Wir versuchen, ihnen diese Arbeit ein Stück weit abzunehmen.“ Dazu füttern sie ihr Programm namens nnU-Net mit bereits markierten Bildern. Daraus lernt es mittels künstlicher Intelligenz – und wendet das Gelernte bei neuen Bildern an.
Das Programm nnU-Net
Der Name nnU-Net ist eine phonetische Abkürzung für „no new net“. Damit möchte das Team um Klaus Maier-Hein und Fabian Isensee ein Zeichen setzen: Für ihr Programm ist kein neues Netzwerk bzw. keine neue Architektur notwendig; stattdessen basiert es auf einer weiterentwickelten Standardmethode.
Mit diesem Ansatz sind Maier-Hein und sein Team nicht allein. Der Einsatz von künstlicher Intelligenz bei der medizinischen Bildgebung ist heute ein stark wachsendes Feld. In vielen großen Krebsbehandlungszentren greifen Strahlentherapeuten bei der Markierung von Tumorgewebe bereits auf Algorithmen zurück. Der Bereich gilt als eines der großen Zukunftsthemen in der Medizin.
Vielseitiger Nutzen ohne aufwändige Programmierarbeit
Die neue Methode nnU-Net der Experten vom DKFZ hat das Forschungsfeld im vergangenen Jahr jedoch gehörig durcheinandergewirbelt – und einen großen Schritt nach vorn gebracht. Das Besondere an dem Programm: es ist nicht für eine bestimmte Segmentierungsaufgabe optimiert, sondern vielseitig einsetzbar – ohne große Programmierarbeit. Und das ist in der Analyse von Bildern mittels KI eine kleine Revolution.
Dass die Heidelberger einen Paradigmenwechsel eingeleitet haben, wurde das erste Mal klar, als sie an einem internationalen Fachwettbewerb für KI-gestützte Bildanalyse teilnahmen, dem sogenannten Medical Segmentation Decathlon. Dort wurde nicht nur ein Datensatz zur Analyse gestellt, sondern gleich zehn verschiedene: Aus der Lunge, vom Gehirn, aus dem Bauchraum und von anderen Körperregionen.
Klaus Maier-Hein leitet die Abteilung Medizinische Bildverarbeitung am Deutschen Krebsforschungszentrum DKFZ. Bild: DKFZ
Schon diese Aufgabenstellung war ungewöhnlich. Denn bisher war es meist so, dass man nur eine einzige Fragestellung hatte. Auf eine solche Anforderung hin haben Medizininformatiker in der Regel einen Algorithmus trainiert und verfeinert. „Man kann sich eine einzelne Bildtyp-Analyseaufgabe so vorstellen wie eine Rennstrecke“, erklärt Klaus Maier-Hein. „Üblich ist es, dass man ein Basisauto hat – das Programm, wie der Algorithmus trainiert wird – und dann mit verschiedenen Anbauten und Tuningmaßnahmen das Auto speziell so verbessert, dass es in bestimmten Kurven und auf bestimmten Teilen der Strecke besser ist, sodass es insgesamt besser wird.“ Bei dem Zehnkampf sind maßgeschneiderte Anbauten allerdings nicht mehr möglich, und so nahmen die meisten Teams einfach mit einem starren Basisalgorithmus teil, der im Schnitt einigermaßen zu den vorgegebenen Daten gepasst hat.
Isensee und seine Kollegen aber wählten einen anderen Weg. „Wir haben das Feintuning des Basisautos optimiert, indem wir es erst systematisiert und dann automatisiert haben.“ Heißt: Das Basisauto der Medizininformatiker schaut sich die Rennstrecke an – und tunt sich darauf ausgerichtet selbst. Nächste Rennstrecke – nächstes, wieder darauf ausgerichtetes automatisches Tuning. Und so weiter.
„Damit sind wir nicht nur auf einer Strecke schneller, sondern überall“, sagt Maier-Hein. Übertragen auf die Analyse von Bildern bedeutet dies: Isensee und Maier-Hein haben zunächst nach Gemeinsamkeiten gesucht, die für alle Bilder gelten. „Wir haben Dinge gefunden, die man bei jedem Bild analysieren kann; das ist zum Beispiel die Pixelkantenlänge. Dazu haben wir dann systematisch Regeln erstellt“, sagt Isensee.
„Auch Medizininformatik-Laien können unser kostenloses Tool unkompliziert anwenden.“
Das neue automatische Tuning überzeugte: Isensee und sein Team gewannen mit Abstand den ersten Platz beim Medical Segmentation Decathlon. Dass sie tatsächlich eine neue Basismethode geschaffen hatten, zeigte sich in den Monaten danach: „Seitdem setzen bis heute bei Segmentierungsaufgaben und Wettbewerben die meisten Teams auf nnU-Net“, sagt Isensee. Das Programm konnte sich in verschiedenen Wettbewerben problemlos gegen spezifisch optimierte Lösungen durchsetzen.
Das kostenlose Open-Source-Programm ist auch von Laien nutzbar
Ein weiterer Vorteil: Auch Medizininformatik-Laien können das Tool unkompliziert anwenden und benötigen vergleichsweise wenig fachliche Unterstützung. Ohne das Programm speziell für die Aufgabe zu verändern und ohne zusätzliches Expertenwissen einfließen zu lassen, erhält man bereits Ergebnisse, die vergleichbar oder sogar besser sind als diejenigen von Algorithmen, die für den spezifischen Einsatz bearbeitet wurden. Für eine breite Anwendung im klinischen Alltag dürfte das in Zukunft ein riesiger Vorteil sein.
Das Team um Klaus Maier-Hein stellt nnU-Net als Open-Source-Tool kostenfrei zum Download zur Verfügung. So ist das Programm sofort einsetzbereit. Aber es gibt auch noch Luft nach oben: Die von nnU-Net vorgeschlagene Konfiguration kann von Experten als Ausgangspunkt für weiteres Tuning benutzt werden. So wurden zum Beispiel im vergangenen Jahr neun von zehn Segmentierungswettbewerbe durch Algorithmen gewonnen, die auf dem Programm basierten – aber noch weiter auf die Aufgabe hin optimiert waren.
Anwendungsmöglichkeiten in der Kernphysik, Weltraum- und Tiefseeforschung
Anwenden lässt sich das Programm übrigens nicht nur in der Medizin. Im Grunde ist jegliche Art von Bildanalyse zu einer guten Qualität möglich. Es muss nur ein Beispieldatensatz vorliegen, der bereits hinzugefügt wurde – damit das Programm davon lernen kann. Ist dies der Fall, könnte nnU-Net künftig zum Beispiel Meeresforschern bei der Analyse von Meeresbodenfotos aus der Tiefsee helfen, in der Kernphysik könnten Aufnahmen mit dem Elektronenmikroskop aufgegliedert werden. Und in der Weltraumforschung könnten zum Beispiel Bilder von der Mondoberfläche untersucht werden. So könnte sich das Programm zu einer Art „KI-Allrounder“ in der Forschung entwickeln.
Leser:innenkommentare